Compartmentalized directed evolution of enzymes using molecular programs as self-selection function
Développement d’une méthode d’évolution dirigée d’enzymes semi in vitro dans des microgouttes à l’aide de programmes moléculaires.
Summary (french below):
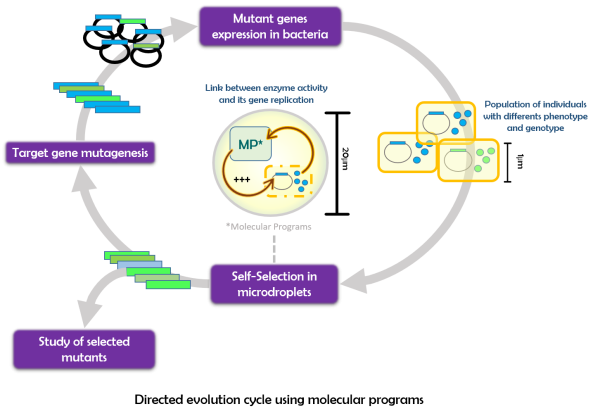
Molecular networks, such as the one using the PEN DNA toolbox (2), are designed to produce short DNA strands interacting within each other’s thanks to a set of 3 different kind of enzymes; a polymerase, a nickase and an exonuclease. Taking short oligonucleotides or enzyme activity at the input, they can generate short oligonucleotides of arbitrary sequence at the output. Enzyme activity can be assessed by these molecular networks thus allowing to produce a correlated amount of primers at the output. These primers can then be used to run the PCR of the enzyme’s gene. Therefore, the artificial network will be able to link the enzymatic activity to a PCR amplification yield. Molecular networks can be modulated by changing their parameters and using them as a selection function can allow to finely tune this step of the directed evolution process. Also, the relative simplicity of molecular programs design and use allows to change easily the selection process parameters at each cycle. Finally, the programs being added externally to the cells enable to test the enzymes in unnatural in vitro conditions.
The number of parallel selection tests that can be run in one evolution cycle is one of the major criteria of efficiency. Microfluidics allows us to generate a large amount of monodisperse water-in-oil microdroplets. Up to 108 parallel self-selections can be run simultaneously on individual copy of the gene with a few amounts of space and reagents. Practically, after generation of a mutant’s library by error prone PCR, bacteria carrying and expressing the mutants are separated in individual droplets containing the molecular program. The process is next initiated in the droplets simply by raising the temperature to at least 45°C. After running the program for some time, each droplet contains an amount of the enzyme’s gene forward and reverse primers corresponding to the activity of the enzyme expressed by the bacterium. A PCR is then launched in the droplets. Its yield in each droplet will then depend on the amount of primers, therefore on the enzyme activity. Droplets are lysed afterwards to retrieve a library enriched in the best mutant genes and a new evolution cycle can be run.
The nicking enzyme Nt.Bst.NBI, one of the nicking enzymes used in the PEN DNA Toolbox, was used as a first model with this adaptive self-selection tool and gives promising results. The several development steps of this new method allowed to optimize bacteria culture conditions and co-encapsulation with the molecular program and PCR reagents. Once the self-selection feedback loop was set up, it was able to successfully select the wild-type gene of Nt.Bst.NBI among a large excess of an inactive version of the enzyme going from an initial faction of 10% to 90% in only one round on selection thus acknowledging for the high selection capacity of the method. Selection cycles on a mutant library of this same enzyme are still running but the analysis of NGS sequencing of the mutants selected in a first cycle already attests an efficient selection process.
(1) F. J. Ghadessy and al. Proc. Natl. Acad. Sci. U.S.A., vol. 98, Apr. 2001.
(2) K. Montagne and al. Mol. Syst Biol., vol. 7, Feb. 2011.
Résumé :
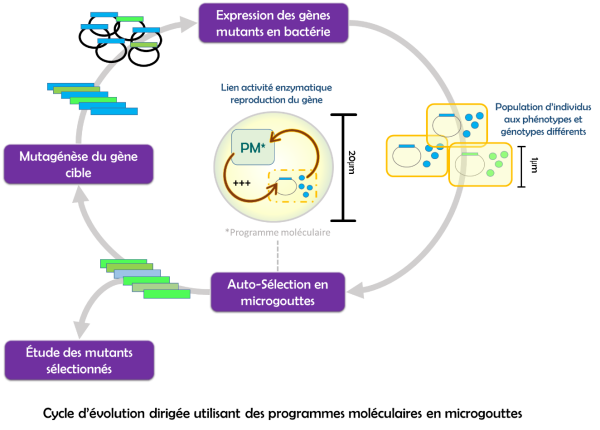
Les programmes moléculaires mis à l’œuvre dans cette méthode d’évolution utilisent la PEN DNA Toolbox (2). Basés sur l’interaction de courts brins d’ADN et d’un ensemble restreint de 3 types d’enzymes (polymérases, exonucléases et nickases) ils permettent de générer de courts oligonucléotides de séquences prédéterminées à partir d’une variété de signaux d’entrée comme un autre brin d’ADN ou une activité enzymatique. Il est donc possible d’utiliser ces programmes moléculaires pour générer des amorces de PCR ciblant le gène à évoluer et ce en quantité dépendante de l’activité enzymatique. De cette manière le rendement en produit de PCR du gène est directement lié à son activité. L’utilisation de programmes moléculaires permet entre autres de pouvoir contrôler cette relation de manière assez précise et d’influencer la fonction de sélection. La relative simplicité de design et d’utilisation des programmes moléculaires permet aussi de pouvoir agir aisément sur la sélection à chaque cycle et de changer les paramètres assez facilement. Enfin le caractère extracellulaire de ces programmes permet de pouvoir tester les enzymes dans des conditions non-naturelles in vitro.
Pour pouvoir réaliser le nombre important de tests requis par les expériences d’évolution dirigée, nous utilisons des gouttes d’eau-dans-l’huile générées par microfluidique afin de créer des compartiments de taille homogène pouvant chacun accueillir un mutant et les réactifs nécessaires. Après génération d’une banque de mutants par PCR error prone, des bactéries contenant et exprimant les mutants sont séparées individuellement dans ces gouttes contenant le programme moléculaire. Le processus est ensuite simplement initié en augmentant la température à 45°C, après un certain temps d’incubation une concentration d’amorces dépendante de l’activité de l’enzyme est atteinte. On lance alors une dont le rendement dépendra de la concentration d’amorces. Après cassage de l’émulsion on retrouve une banque de mutants enrichies avec les meilleurs gènes qui sera utilisée pour créer une nouvelle banque et répéter les cycles d’évolution.
L’enzyme étudiée comme premier modèle de cette méthode d’auto-sélection est une des nickases utilisées dans les programmes moléculaires de la PEN DNA Toolbox : Nt.Bst.NBI. Les étapes de développement de cette nouvelle méthode ont permis d’optimiser les conditions de cultures et de co-encapsulation des bactéries avec les réactifs nécessaires à l’étape de programmation moléculaire et de PCR. Elles ont rendu possible la sélection du gène wild-type de Nt.Bst.NBI parmi une large majorité de mutants inactifs passant d’une fraction initiale de 10% à une fraction finale de 90% en un seul cycle démontrant ainsi la forte capacité de sélection de la méthode. Les cycles de sélection sur une banque de mutant de cette même enzyme sont en cours et l’analyse des résultats de séquençage NGS des mutants sélectionnés dans un tout premier cycle montre déjà les signes d’une sélection efficace.
(1) F. J. Ghadessy and al. Proc. Natl. Acad. Sci. U.S.A., vol. 98, Apr. 2001.
(2) K. Montagne and al. Mol. Syst Biol., vol. 7, Feb. 2011.