Programmer des billes avec de l’ADN ? Cela peut paraître incroyable mais c’est pourtant le défi réalisé par l’équipe de Yannick Rondelez et son équipe, initialement au LIMMS (CNRS, Université de Tokyo) puis au sein du laboratoire Gulliver à l’ESPCI Paris. La méthode est assez simple : implanter des séquences d’ADN bien spécifiques à la surface de microparticules, elles même baignées dans un mélange contenant des enzymes. Les séquences, encodant la fonction de chaque particule, interagissent avec les enzymes pour produire des brins d’ADN qui permettent la communication entre les particules, générant des comportements collectifs à l’échelle de populations de particules. Ces travaux, publiés dans la revue Nature Nanotechnology, sont une première percée vers le développement de calculateurs biologiquement inspirés complexes, ou de nouveaux systèmes de diagnostic médicaux.
L’ADN comme logiciel, un mélange d’enzymes comme processeur, c’est l’approche proposée par Yannick Rondelez et son équipe pour développer des programmes moléculaires capables de manipuler de l’information. A l’instar du code binaire (0 et 1) en informatique, il est possible d’utiliser le code génétique (A, C, G et T) pour encoder des informations, ici dans des séquences d’ADN synthétiques. Le mélange enzymatique interprète ces séquences nucléiques et synthétise les séquences correspondantes, permettant de réaliser diverses opérations élémentaires (activation, inhibition …). Ces opérations sont alors connectées en circuits moléculaires artificiels permettant de réaliser des calculs, de stocker de l’information ou de générer des comportements dynamiques (oscillation, multistabilité…).
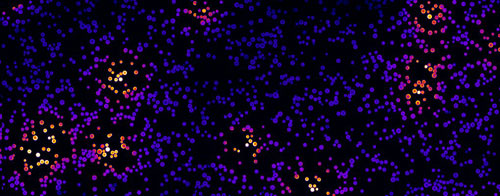
Les chercheurs de l’équipe de Gulliver ont démontré la possibilité de programmer des microparticules (env. 30 µm) avec ces circuits moléculaires de manière à attribuer une fonction spécifique à chacune d’entre elles. En contact avec une solution enzymatique, la machinerie ADN induit la production de brins d’ADN en réponse à un stimulus donné. Par diffusion de ces brins, les particules peuvent communiquer et émettre à leur tour des séquences. Il devient possible de programmer des milliers de particules de sorte que ces populations adoptent collectivement des comportements dynamiques.
Fronts d’activation
Dans un premier type d’expériences, des populations de particules autocatalytiques (les séquences qu’elles portent encodent la réplication d’un brin d’ADN donné) sont immergées dans la solution enzymatique. Parmi elles, certaines sont programmées pour amplifier spontanément ces séquences qui diffusent librement, déclenchant la production de ces mêmes brins par les billes voisines. De proche en proche, le signal se propage et active séquentiellement toute la population.
Dans un système employant 4 types de particules, les chercheurs ont développé un « sonar biochimique » capable de calculer la distance entre une particule émettrice et une particule réceptrice. L’émetteur envoie un signal qui traverse une première population de particules. Lorsque ce signal atteint le récepteur, celui-ci le convertit en un signal orthogonal (une séquence d’ADN différente), qui retourne jusqu’à l’émetteur via une seconde population de particules.
Emergence de colonies
Ici, deux populations de particules encodent des règles réciproques. L’une produit le substrat de l’autre et inversement. Ainsi des particules de type opposé doivent interagir synergiquement afin de transiter vers l’état actif. Les chercheurs peuvent alors observer à l’échelle microscopique l’émergence de colonies (par analogie aux colonies bactériennes) due à l’amplification localisée autour de particules synergiques voisines.
A terme les chercheurs espèrent utiliser ces particules programmables pour la détection ultrasensible de marqueurs biologiques comme les microARN. En effet, ces biomarqueurs, détectables à partir d’un simple prélèvement sanguin, sont caractéristiques de certains cancers et autres pathologies. Ces nouvelles approches de médecine « moléculaire » permettraient un meilleur suivi des patients, et un diagnostic précoce et non invasif de certaines maladies pour lesquelles les examens peuvent être lourds. Un brevet a été déposé sur le procédé utilisé pour déposer un « programme » sur une particule, associé à son système de détection.
Publication associée :
Microscopic agents programmed by DNA circuits
Gines et al., Nature Nanotechnology
doi:10.1038/nnano.2016.299








